6.5 降维分析 EMP_dimension_analysis
模块EMP_dimension_analysis支持多维数据的降维分析,包含主成分分析PCA、主坐标分析PCoA、偏最小二乘回归PLS,正交偏最小二乘回归OPLS和umap等方法。
6.5.1 微生物数据的降维(也称β-多样性)
注意:
如果需要PCA降维,只需要指定参数
如果需要PCA降维,只需要指定参数
method为pca即可
🏷️示例:
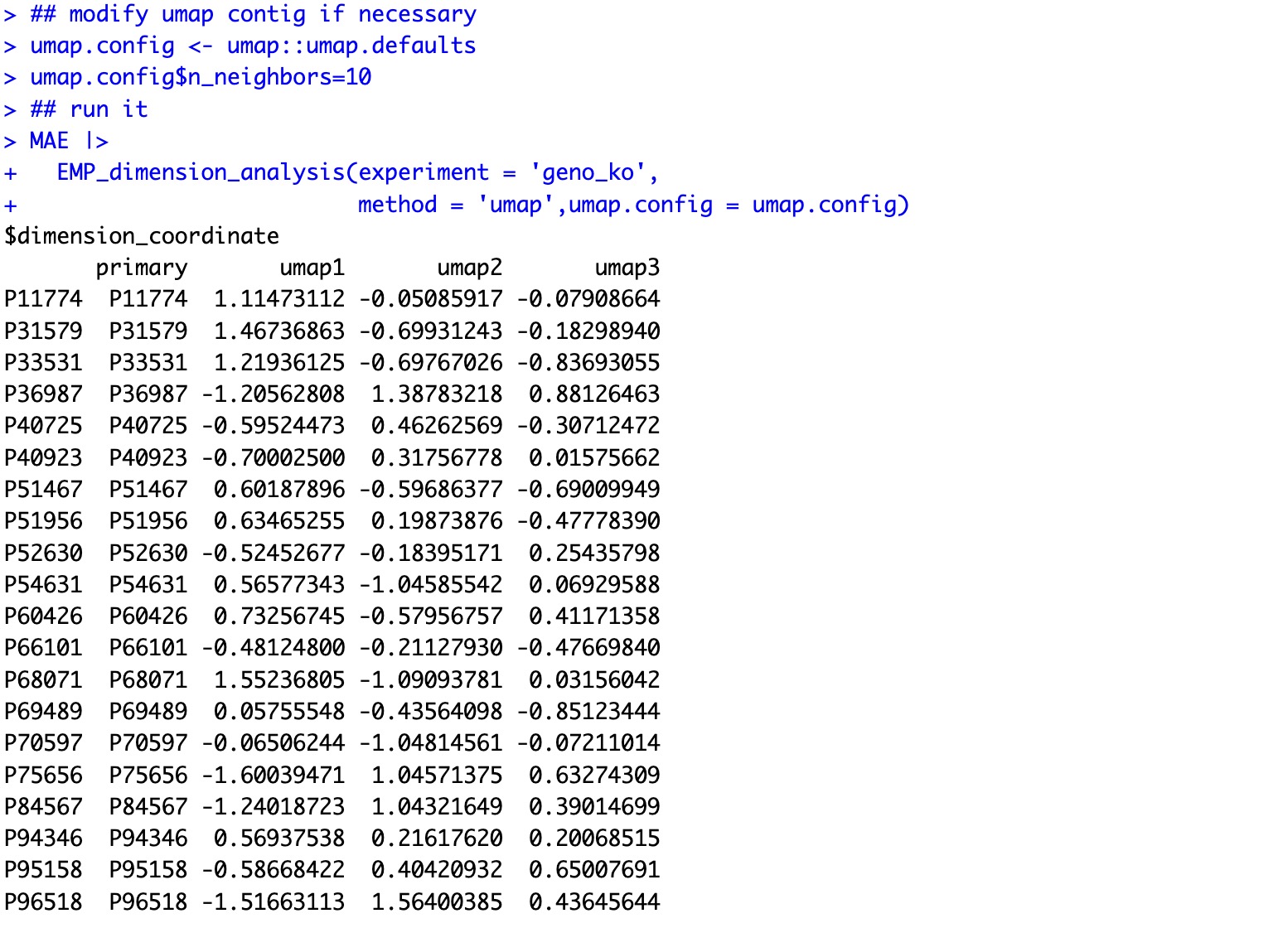
从MAE对象中提取组学项目taxonomy的assay,利用EMP_identify_assay筛选核心微生物,利用EMP_collapse将核心微生物折叠为属级别数据,利用EMP_dimension_analysis进行PCoA降维分析。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_identify_assay(method='default',estimate_group = 'Group') |>
EMP_collapse(estimate_group = 'Genus',collapse_by = 'row') |>
EMP_dimension_analysis(method = 'pcoa',distance='bray')
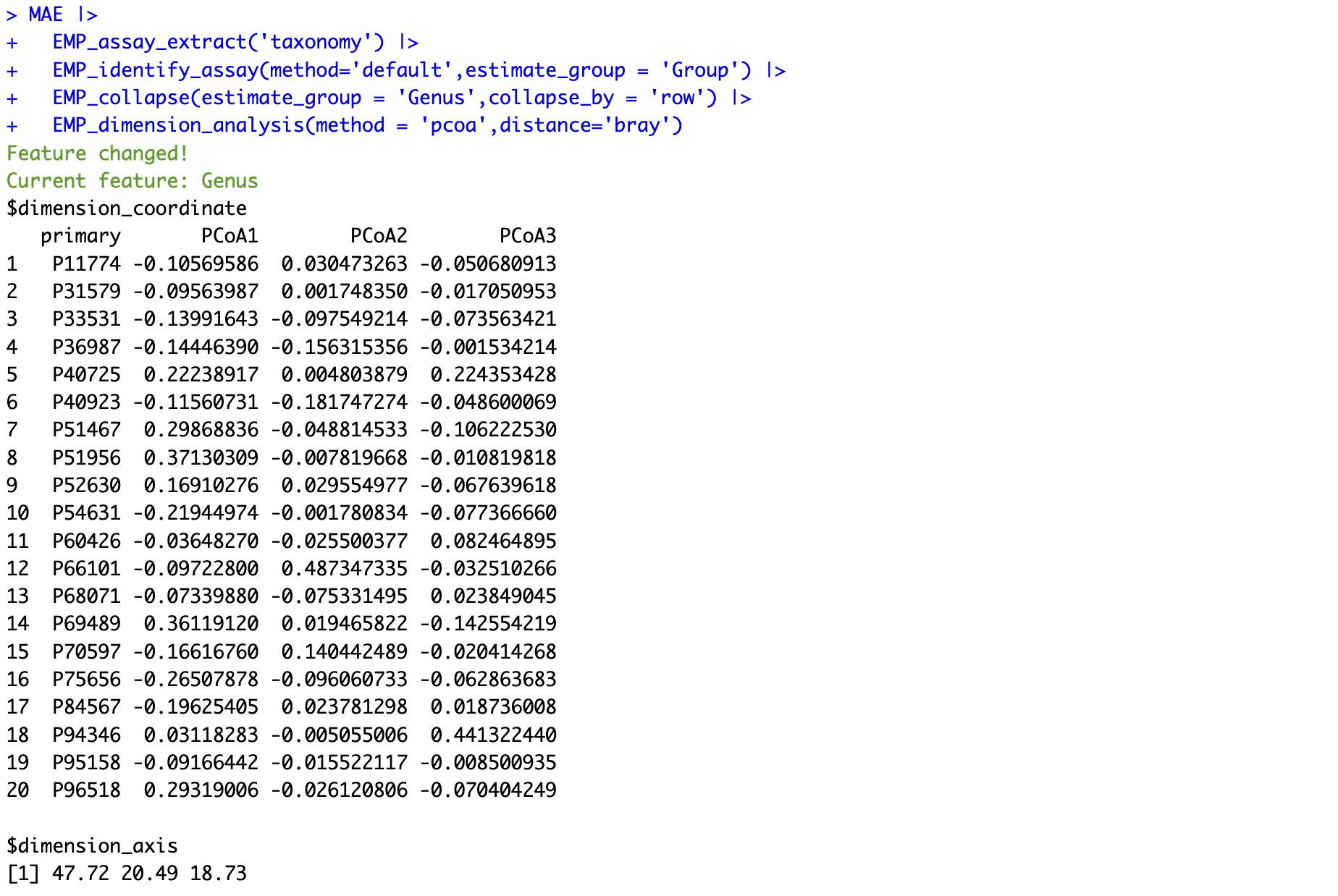
降维分析的结果可以使用EMP_scatterplot进行可视化。
注意:
参数
参数
show支持从p12、p13、p23三个维度呈现降维结果,并提供html交互式图形。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_identify_assay(method='default',estimate_group = 'Group') |>
EMP_collapse(estimate_group = 'Genus',collapse_by = 'row') |>
EMP_dimension_analysis(method = 'pcoa',distance='bray') |>
EMP_scatterplot(estimate_group='Group',show='p12html',ellipse=0.3)
6.5.2 代谢组学数据PLS/OPLS降维分析和特征代谢产物筛选
注意:
如果需要OPLS模型,只需要指定参数
如果需要OPLS模型,只需要指定参数
method为opls即可
🏷️示例:
从MAE对象中提取组学项目untarget_metabol的assay,根据二级代谢产物KEGG注释折叠数据并进行相对丰度转化,进行PLS降维分析、获取代谢产物的降维坐标及VIP值,根据VIP值筛选特征代谢产物。
MAE |>
EMP_assay_extract(experiment = 'untarget_metabol') |>
EMP_collapse(estimate_group = 'MS2kegg',collapse_by='row',
na_string = c("NA", "null", "","-"),
method = 'mean',collapse_sep = '+') |>
EMP_decostand(method = 'relative') |>
EMP_dimension_analysis(method = 'pls',estimate_group = 'Group') |>
EMP_filter(feature_condition = VIP >2)

6.5.3 KO注释数据的UMAP降维分析
🏷️示例1: 使用UMAP算法进行数据降维
MAE |>
EMP_dimension_analysis(experiment = 'geno_ko',
method = 'umap')
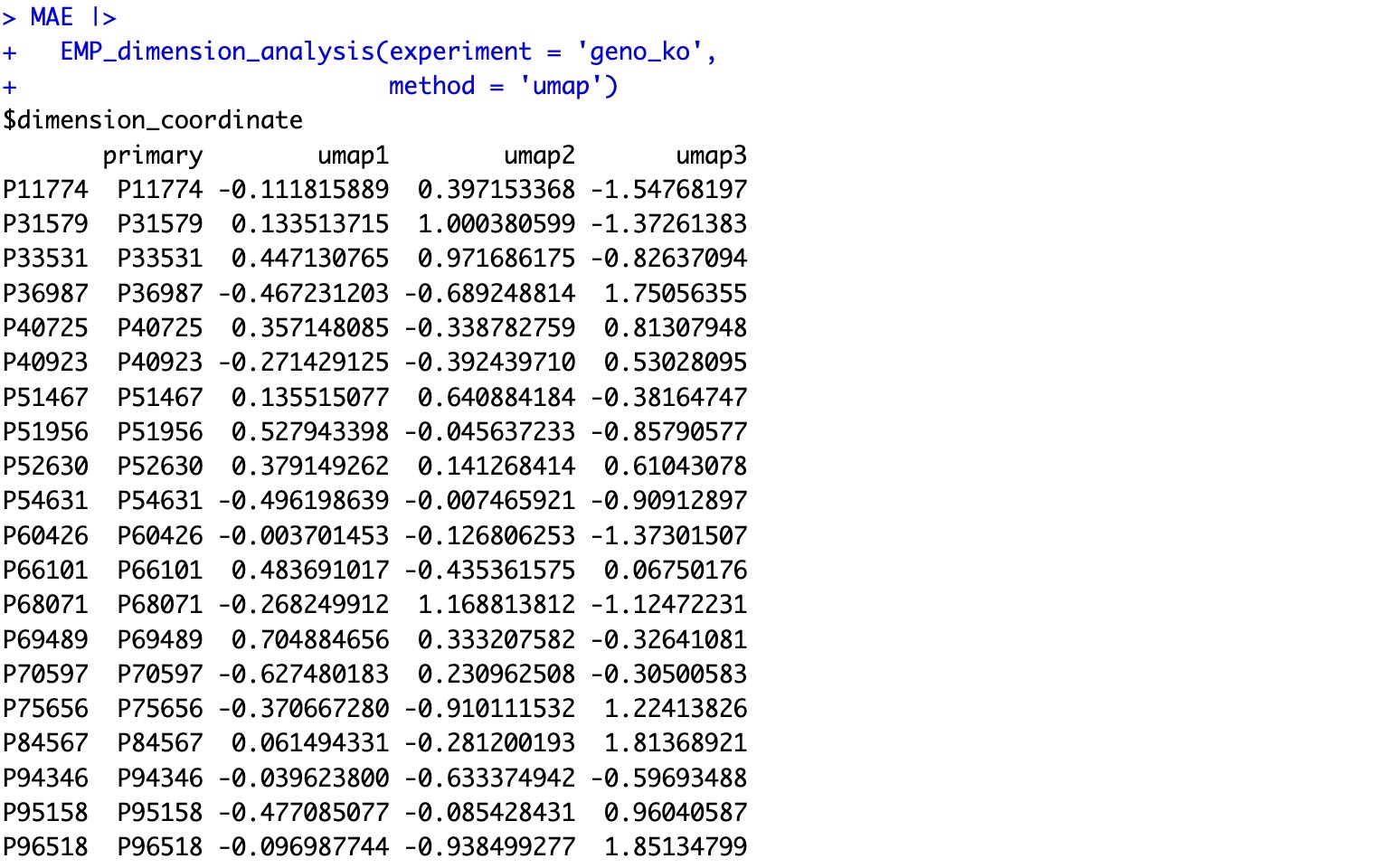
🏷️示例2: 修改UMAP算法运行参数
从 umap包内获得参数文件
umap.config <- umap::umap.defaults
umap.config$n_neighbors <- 10
在模块函数中输入参数设置文件
MAE |>
EMP_dimension_analysis(experiment = 'geno_ko',
method = 'umap', umap.config = umap.config)